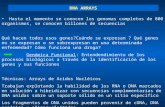
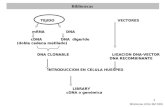
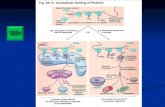
BM-3.Empaquetament Del DNA
-
Upload
ignasigrasscosta -
Category
Documents
-
view
229 -
download
0
description
Transcript of BM-3.Empaquetament Del DNA

Tema 6: Empaquetament del DNA
1. Empaquetament del genoma en virus i procariotes
Com ja sabem els virus estan formats per càpsides que englovan el material genètic. Aquesta capside te un espai reduït i per tant el genoma a part de ser reduït de per si també haurà de presentar mecanismes per empaquetar-‐se
1.1 Virus del mosaic del tabac Càpsida: Presenta una simetria helicoïdal i esta formada per una proteïna que es troba repetida n vegades. Aquesta proteïna presenta una forma triangular i te la capacitat d’associar-‐se amb el RNA, per tant la càpsida es formarà al voltant del material genètic i permetrà que aquest s’empaqueti Segons les condicions del medi de pH i força iònica la proteïna pot estar sola, formant petits agregats, discs plans o helicoides o directament una hèlix. La hèlix es formar aper la unió de diversos helicoides i posteriorment s ‘ompliran els forats que quedin. Plegament del DNA: Si observem amb una fotogrfia podem veure que mentre es forma la càpsida els dos extrems del RNA es comporta de manera diferent. Ja que en un inici es veu una càpsida molt petita amb poc RNA associat i el extrem 3’ serà mes llarg que el 5’. Mentre es va formant la càpsida el extrem 3’ s’anirà escurçant mentre que el curt es mantindrà amb la mateixa longitud. Aquest fet es degut a que el extrem 3’ s’anirà enrotllant gracies a la càpsida. Finalment el extrem 5’ també entrarà.
1.2 Bacteriofag Càpsida: Presenten un simetria complexa. En aquest cas primer es formarà la càpsida icosaèdrica, permetrà l’entrada del DNA i per tant el seu empaquetament, i posteriorment s’unirà amb la part helicoïdal. Plegament del DNA: En aquest cas com ja hem dit tindrem la capside icosaèdrica formada. Aquesta presenta un complex proteic especial que es capaç d’internalitzar la molècula de DNA mitjançant canvis de conformació provocats per la hidròlisi de l’ATP que permeten que tot el complex vagi girant i que per tant baixi entrant el DNA. Es a dir funciona com un tornillo (DNA) i una femella (complex proteic) S’han realitzats estudis de nanotecnologies que hem permès estudiar aquest mecanisme d’empaquetament. El experiment utilitzava pinses òptiques en les quals un extrem de la pinsà presentava unit la càpsida icosaèdrica i l’altre una molècula de DNA mitjançant biotina-‐estreptovidina. D’aquesta manera s’ha pogut mesurar la força de tracció que fa el complex proteic sobre el DNA i també la cinètica de l’entrada del DNA
JR.Daban BIOLOGIA MOLECULAR Figura 6.4
-------------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.4
-------------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.5
-------------------------------------------------------------------------------------------------

sent que aquesta va disminuït com més DNA ja es troba en el interior fins que la velocitat es 0.
2. Empaquetament del DNA en procariotes Les cèl·∙lules eucariotes no presenten nucli però també han de tenir el seu DNA empaquetat ja que sinó no hi cabria en el interior de la cèl·∙lula. Empaquetament: El empaquetament te lloc en el nucleoide i en dos nivells:
• Proteines: S’anomenen histone-‐like que tenen la propietat d’unir-‐se al DNA i provoca que aquest s’enrotlli al seu voltat.
• Llaçades: Worcel a més a més va proposar que DNA de les procariotes estava formant una mena de llaçades. Aquesta conclusió la va extrapolar quan va fer estudis de sedimentació del DNA de E.coli i va veure que presentava un coeficient de sedimentació molt gran (1500). Aquest coeficient era molt mes gran del que s’esperava i l’única raó de ser era perquè el DNA tenia que estar empaquetat. A continuació va tractar el DNA amb DNAasa 1 que provoca nikacio del DNA i va veure que el coeficient de sedimentació disminuïa, però en canvi quan el tractava amb el BrEt que s’intercalava amb el DNA va veure que el coeficient de sedimentació en primer lloc disminuïa i despres tornava a augmentar. Per tant aquest DNA havia de presentar una estructura de plegament que presentes topologia, com la llaçada, ja que la nikació la relaxava i la BrEt el relaxava primer però despres el eia enrotllar cap al altre canto.
3. Empaquetament del DNA en el nucli. Cromatida Cromatina: Pels químics es tot aquell material que podem extreure del nucli d’una cèl·∙lula eucariota i pels histològics es tot allò que es tenyeix amb un colorant bàsica en el nucli. La cromatina dons no es DNA sol sinó que també inclou:
• RNA
JR.Daban BIOLOGIA MOLECULAR Figura 6.1
-------------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.2
-------------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.3
-------------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.6
-------------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.7
-------------------------------------------------------------------------------------------------

• Proteïnes no histoniques que depenen del estat metabòlic • Histones: Les histones es troben en la mateixa proporció que el DNA i això es
perquè es troben sempre associat en ell. Per tant en el nucli el DNA mai esta sol sinó que sempre esta unit a histones.
3.Histones Definició: Son unes proteïnes que poden associar-‐se el DNA. Aquesta associació es pot fer la presencia d’una gran quantitat d’aa carregats positivament com poden ser l’Arg i la Lys que li permeten fer interaccions electrostàtiques amb el DNA. D’aquesta manera quan afegim una concentració 2M de NaCl capaç d’apantallar les carregues les H es dissocien amb el DNA Tipus: Existeixen 5 tipus de histones: H1, H2A, H2B, H3 i H4. D’aquestes podem veure com la H1 presenta una massa molecular el doble que totes les altres i presenta una gran quantitat de Lys en comparació amb la de Arg, mentre que totes les altres presenten un pes molecular molt semblant i una proporció de Arg i Lys semblant. La sequencia d’aa esta molt conservada en totes les (H3 i H4 les que més) menys en la H1, i actualment es la proteïna mes conservada que coneixem. Aquest fet demostra que tots els aa de la seqüencia de les H son molt importants per tal de realitzar la seva funció estructural en el DNA i que un sol canvi fa que el individu no pugui prosperar. Ex: Entre una seqüencia de H4 de vedella i de pèsol nomeshi ha un canvi de tipus conservatiu entre una Ile per una Arg
3.1 Estructura de les histones Proteolisi: Es va realitzar típic assaig estructural de proteïnes com pot ser la proteòlisis suau. Els resultats van ser que existien zones que es degradaven i per tant no estaven protegides però d’altres que si que es degradaven i per tant tenien que estar protegide. A més a més van veure que les zones que es degradaven eren riques en Lys i Arg i les que no es degradaven eren riques an aa apolars. Estructura: A partir dels resultat anteriors podem dir que les H presenten una estructura formada per una forma globular (�)formada per residus apolars i una forma no globular (+) formada per residus de Arg i Lys principalment. En el cas de la histona H1 aquesta regió no globular es troba en els dos extrems de la seqüencia i per tant diem que presenta una cua N-‐ter i una cua C-‐ter (seran les zones mes variables en seqüencia). En canvi la resta de histones nomes presenta una regió no globular en el extrem N-‐ter i per aquest motiu direm que presenta una cua N-‐ter.
JR.Daban BIOLOGIA MOLECULAR Figura 6.9
-------------------------------------------------------------------------------------------------

3.2 Modificacions postradiccionals en les H Tipus: Existeixen diferents tipus de modificacions i cada una d’elles te un efecte diferent
• Metilació: es produexen en les Lys i son capaços d’apantellar la carrega per no eliminar-‐la i per tant tenen un efecte neutre. Poden arribar ha haver-‐hi fins a 3 metilacions
• Fosforilació: Es produeix en les Ser i les Thr. Es una carrega negativa que provoca canvis en la interacció entre H i DNA i per tant canvis en l’empaquetament
• Acetilació: Es produix en Lys. Son capaços d’eliminar una carrega negativa i per tant provoca canvis en la interacció entre H i DNA i per tant canvis en l’empaquetament. D’aquesta manera es pot regular l’expressió gènica i podem afiramar que
o Desasetilases: Provoquen la desacetilacio de les Lys, conseqüentment es guanya una carrega positiva i per tant empaqueta mes el DNA. Com a conseqüència hi ha una repressió de l’expressió gènica. Per tant son aquets enzims seran coneguts com repressors
o Acetilacio Provoquen la acetilació de les Lys, conseqüentment es perd una carrega positiva i per tant desempaqueta mes el DNA. Com a conseqüència hi ha una activació de l’expressió gènica. Per tant son aquets enzims seran coneguts com coactivadors
Localització: Les modificacions postraduccionals sempre es duen a terme en les cues
4. Nucleosoma
4.1 Primers descripció del nuclosoma Definició: Es va descriure que la presenta una certa estructura basada en una seria de repeticions unides per un fragment de DNA d’uns 200pb Hewish i Burgonye: Eren dos científics australians que estaven investigant nucleases endògenes, i específicament amb endonucleases de fetge. Una de les digestions que van fer sobre cromatina la van separar amb un gel electroforètic i van observar un patró de bandes repetiu que tenia que ser resultat d’una col·∙lecció de fragments amb longituds constants. Aquest patró no era esperat ja que si es una nucleasa endogen el que caldria esperar es un conjunt de fragments tots ells amb longituds diferents i per tant que es crees una llapisada. Val a dir que aquest patró ja va estar descrit per altres científica però mai ningú s’havia preguntat perquè passava. La seva hipòtesis es va basar en que la cromatina tenia que tenir una estructura senzilla que es bases en un conjunt de subunitats repetides i seperades entre elles per un espai físic suficient perquè la nucleasa pogués fer la digestió. Segons l’anàlisi del gel, l’espai físic havia de ser de 200pb. Cal remarcar que no treballàvem amb la nucleasa microcoal
JR.Daban BIOLOGIA MOLECULAR Figura 6.9
-------------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.9
-------------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.9
-------------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.10
-------------------------------------------------------------------------------------------------

4.2 Estudis de microscòpia òptica Olins&Olins: Van realitzar un xoc osmòtic a les cèl·∙lules, es a dir, les van col·∙locar en un medi sense sal (aigua pura) i com a conseqüència tant les cèl·∙lules com els nuclis van rebentar. El material del nucli que van obtenir el van observar per microscòpia electrònica i van poder veure que existien una meda de boletes que presentaven uns 10nm al qual els i envoltava i unia una mena de fil que podia ser el DNA. Hem de remarcar que aquesta estructura no es pot estudiar per difracció de raig X (actualment nomes s’ha pogut resoldre una part del nucleosoma) i per tant hem d’utilitzar tècniques estructurals indirectes. Per aquest motiu l’empaquetament del DNA ha sigut un tema molt important en la BM ja que es requereix d’imaginació i de moltes tècniques diferents.
4.3 Estudis del complex de Histones del nuclosoma Definició: Les histones nucleosomiques, es a dir, H2A,H2B, H3 i H4, en condicions fisiològiques tenen la capacitat de formar un octàmer. Aquest octàmer esta format per:
• Tetramer de H3 i H4 • Dos dímers de H2A i H2B
Kornenberg: Va estudiar el comportament del les histones nucleosomiques i va veure que quan aquestes no es trobaven en condicions fisiològiques formaven un octàmer a partir de dos copies de cada histona. Per analitzar be aquest octàmer va col·∙locar les H en condicions fisiològiques (0,2M NaCl) ja que quan feia una gel filtració (Sephadex G100) i després mirava l’absorbància veia com es formaven dos pics diferents un d’ells format per H3H4 i l’altre a H2AH2B malgrat que totes les H presentaven un pes molecular igual. La hipòtesis que va crear va ser que H3H4 generava un tetràmer mentre que H2AH2B formava dímers. Aquesta hipòtesis va estar demostrada quan es va fer un estudi d’entrecreuament químic (croos-‐link) mitjançant dimetilsuberimidat. Aquesta molecula era capaç d’establir un enllaç en els seus dos extrems amb dos amines diferents, per tant es capaç d’unir de forma covalent dos proteïnes que estan unides de forma no covalent. Van afegir aquest reactiu en una solució d’H3 i H4 nucleosomiques en codicions fisiològiques i amb una concentració suficient perquè es crees una col·∙lació entre H3 i H4 no unides i H3H4 unides en les diferents formes o combinacions possibles. i posteriorment van fer un gel electroforètic amb SDS (desnaturalitzant) i els resultats van demostrar que es van formar monòmers, dímers, trímers, i tetràmers. Quan es va repetir el procés amb l’H2A i H2B nomes es van veure dímers i monòmers. La rao per la qual quan les concentracions salines son elevades es forma un octàmer segurament es deuen a que d’aquesta manera les H poden neutralitzar millor les carregues que hi han en el medi. Això mateix es el que deu pasar quan una H es troba amb el DNA ja que aquest te carregues negatives igual que el Cl
JR.Daban BIOLOGIA MOLECULAR Figura 6.10
-------------------------------------------------------------------------------------------------

4.4 Model estructural del nucleosoma Definició: Va ser un model proposat per Kornenberg a partir de tota la informació que es tenia del nucleosoma fins al 1974. Descriu que el nuclosoma es un compost format per 200pb que conte un octàmer d’H format per 2 parelles de H2A, H2B, H3 i H4 i una sola subunitat de H1 (en alguns teixits es diu H5) Malgrat no va estar definit per Kornenberg hem de definir que el nucleosoma esta format per la partícula nucli i el DNA-‐linker. LA partícula nucli esta formada pel octàmer d’H i 146pb i el DNA-‐linker esta entre mig de dos octàmers d’H i esta formada per 60pb
4.5 Estudis de la situació del DNA en la partícula nucli Definició: El DNA es troba per fora del octàmer d’H. Esta formant dos voltes Noll: Es va tractar la cromatina amb DNAasa 1 i va fer un gel electroforètic desnaturalitzant. En aquest es van veure un patró de bandes separades entre elles per 10pb (una volta de hèlix) al llarg de les 146pb que formava el nucleosoma, per tant, e DNA nomes pot estar per fora ja que si esta per dintre no veuriam cap tall, i si estigues mig a mig a fora hi hauria una zona sense talls Luther: Es va tractar nucleosomes que presentaven DNA amb el seu extrem 5’ marcat radioactivamen amb DNAsa 1 durant un temps prolongat i posteriorment va fer una electroforesis amb un gel desnaturalitzant i una autoradiografia. En el els gels es podia veure moltes bandes i o llapisades però en canvi hi havia zones en les quals no hi havia cap banda independentment del temps d’exposició amb la DNAasa1, per tant aquestes zones eren inaccessibles. Aquets llocs eren el 30, 60, 80, 110 i 140 en els dos cantons Mitjançant el estudi de molts gels electroforètics diferents i aplicant matemàtiques va poder determinar quines eren les zones de tall mes probable. Va representar les probabilitats de tall al llarg de les 140pb de la partícula i va veure que existia un eix de simetria rotacional binari amb un interval entre els iguals de 80.. Per tant si hi havia una eix de simetria rotacional binaria degut a la protecció que feia el nucleosoma, aquest tenia que presentar una estructura acorde amb això.
JR.Daban BIOLOGIA MOLECULAR Figura 6.11
-------------------------------------------------------------------------------------------------JR.Daban BIOLOGIA MOLECULAR Figura 6.11
-------------------------------------------------------------------------------------------------

La conclusió va ser que el DNA feia una volta i 3/4 per fora del nucleosoma i que cada volta ocupava 80 pb d’aquesta manera la simetria rotacional binaria es creava perquè els iguals estaven un a sobre dels altres
4.5 Primers cristalls de nucleosoma Klug: Eren uns cristalls de molt baixa resolució però que combinats amb altres resultats obtinguts per altre gent que va utilitzar difreccio de neutros va permetre confirmar l’estructura descrita per Luther. La difracció de neutrons presenta la capacitat de seleccionar si es vol veure la proteïna o el DNA depenen del medi. Es va poder veure que la helicoide del DNA presentava un diàmetre més gran que no pas el que feia la proteïna i que aquest donava 1 volta i 3/4 al seu voltant i que el gir era levogir. També va poder determinar que l’alçada del octàmer era de 6nm i el diàmetre era de 11nm i si contem el DNA serà de 13nm
4.6 Topologia Funció dels nucleosomes en topologia: Es va fer un experiment en el qual es van utilitzar una col·∙lecció de solucions de DNA circular covalentment tancat amb una concentració creixent de H i una col·∙lecció idèntica però a la qual hi havíem afegit la topoisomerasa I (ells l’hi diuen untwiting extract). A continuacio fem una electroforesis amb un gel d’agarosa i podem veure que en les carrils de les solucions sense topo (n) que efectivament les H son capaces de produir-‐me un enrotllament que fa que el DNA migri mes, però si mirem els carrils de les solucions amb topo podem veure que els
JR.Daban BIOLOGIA MOLECULAR Figura 6.12
-------------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.12
-------------------------------------------------------------------------------------------------JR.Daban BIOLOGIA MOLECULAR Figura 6.12
-------------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.13
------------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.13
------------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.13
------------------------------------------------------------------------------------------------

carrils que tenen menys histona (a) la topo es capaç de relaxar però els carrils que tenen mes histona la topo no es capaç de relaxar. Això es degut a que quan el DNA s’enrotlla en la partícula nucli fa uns girs capa a l’esquerra que han de ser compensats per uns girs a la dreta en les zones que no hi ha histones i la relaxació de la topo I nomes es pot fer per zones del DNA en les quals no hi ha histones i per tant quan la concentració d’H es elevada no es podrà produir la relaxació. Paradoxa del valor de L en el nuclosoma: Com ja hem vist en la partícula nucli es creen dos voltes tiroïdals levogires i per tant això crea un W=-‐2 i faria que el DNA no associat a H tingues un valor de W=+2. Posteriorment van analitzar la topologia i per fer-‐nos una idea del procediemnt hem de imaginar un DNA circular relaxat amb un valor de L=20 i un valor de T=20 i per tant W=0. A continuació es col·∙loca un sol ocamer d’H i això crea que el DNA es superentrolli. Poseteriorment aplcas una topo I que es capaç de relaxar el DNA que es troba sense H i finalment eleimnas el nucleosoma i com a resultat obtens un DNA amb un valor L=19 degut a que ΔW ara es de -‐1, i no pas ΔW=-‐2 que es el que caldria esperar. Això va generar el que es coneix com a Paradoxa del valor de L en el nuclosoma. Per tal de poder trobar una solució es va proposar que realment ΔW=-‐2 i que per tant si ΔL=-‐1 ΔT=+1 per tant el nuclosoma ha de ser capaç de fer variar el pb/volta de 10,5 a 10, es a dir fer variar l’estructura secundaria del DNA ja que: El motiu pel qual el nucleosoma provoqui un ΔL=-‐1 es segurament degut a que d’aquesta manera no es produeix un superenrotllament tant pronunciat que podria tenir associat problemes topològics.
4.7 Plegament del DNA en el nucleosoma 4.7.1 Seqüencia de plegament Definició: El DNA ha de fer un gran corbament quan s’uneix a les histones ja que dona dos voltes en 146pb. Aquest corbament tant pronunciat provoca que les bases del que
JR.Daban BIOLOGIA MOLECULAR Figura 6.14
------------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.14
------------------------------------------------------------------------------------------------

estan tocant la histona presentin una distancia entre bases de 0,3, mentre que les que es troben en el exterior presenten una distancia entre bases de 0,4. La distancia recordem que ha de ser de 0,34, per tant cap dels dos esta estable. Per tant es necessari esbrinar si hi ha alguna preferència en la estructura en la seqüencia del DNA que ficiliti més o menys el plegament Travers: Va analitzar la seqüencia de DNA de 187 nucleosomes agafats a l’atzar i va determinar quines probabilitats hi havia que un nucleòsid o grup de nuclosids es trobes en cada posició. El que va obtenir era un gràfic de 146pb que presentava un altre cop un eix de simetria. Es representaven per una banda la sequencia GC i per l’altra al TTAA i es veia una distribució sinosoidal en que la longitud entre els pics era de 10pb i que els dos gràfics estaven en antifase. Per tant va determinar que qualsevol seqüencia es capaç de formar un nucleosoma pero que el nucleosoma atacarà el DNA per aquella posició en la qual en el interior del corbament i trobem T i A i en el exterior G i C. Per tant podem dir que la histona te preferències anisotropiqes
4.7.2 Repulsions en el plegament Es coneix que el octàmer tenen una carrega global de 142(+) i en canvi el DNA te una carrega global de 146(-‐)·∙2=292(-‐). Per tant la H apantalla nomes la mitat de les carregues però en canvi aquest corbament permet que les carregues de la cara que no esta en contacte amb el octàmer i que per tant no estan apantallades estiguin mes separades entre si, conseqüentment hi ha menys repulsió i això fa que el plegament es pugui veure facilitat.
4.8 Disposició de les H en el nucleosoma Definició: Es basa en conèixer quines H interaccionen amb el DNA i com aquestes es disposen en l’octàmer d’H. El problema però es que fins fa molt poc no es va poder cristal·∙litzar el nucleosoma. Mirzabekov: Utilitza un reactiu anomenat Me2SO4 que era capaç de fer un enllaç covalent amb la His i posteriorment provoca el trencamentr del enllaç fosfodièster en la cadena de DNA just per la base a la qual s’ha unit.
JR.Daban BIOLOGIA MOLECULAR Figura 6.15
------------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.15
------------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.16
------------------------------------------------------------------------------------------------

Va marcar radioactivament els extrems 5’ i a continuació va col·∙locar aquest reactiu en una solució que contenia DNA unit a histones amb una concentració i un temps de reacció que com a resultat va obtenir una col·∙lecció de fragments diferents units a les diferents H del nuclosoma (un sense cap unió, un amb unió a H3, un amb unió a H4..) A continuació va realitzar una electroforesis bidimensional en la qual:
• Primera dimensió: Es capaç de separar segons la mida diferents cadenes de DNA unides a les histones. Per tant els que corrin mes serà aquells que presenten un fragment de DNA més curt i els que corrin menys seran els que presenten un fragment de DNA més llarg. Hem de tenir en compte però que depenen de la H a la que va unida dos fragment de la mateixa mida pot corre mes o menys pero la diferencia es tant petita que la considerem negligible
• Segona dimensió: Apliquem un tractament capaç d’eliminar el DNA i tornem a separar segons la mida. Per tant podrem separar les diferents histones ja que cadascuna presenta una massa molecular diferent i per tant de menys migració tenim H3, H2A, H2B i H4
Posteriorment fem una autoradiografia. Analitzant els resultats va poder fer un mapa de la posicio de les histones respecte a la seva interacció amb el DNA. Lògicament te mapa també presenta una simetria i on es veu que una mateixa histona pot interaccionar a diferents llocs. Podem veure que hi ha zonen en les qusl no hi ha intereccio i aquest fet es logic ja que realment el DNA te una estrucctura tridimensional que imposibilita que la totalitat cara interna del DNA intereccioni amb la H
4.9 Resolució de l’estructura de l’octamer Aron Klug: Va aconseguir cocristalls de octàmers de H sense DNA a partir de crear solucions de H a molt alta concentració fins al punt de formar agragats. El resultat el va observar per microscòpia electrònica i va determinar els següents paràmetres:
• La histona presenta una estructura en falca • Presenta una simetria binaria
JR.Daban BIOLOGIA MOLECULAR Figura 6.17
------------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.18
------------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.18
------------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.18
------------------------------------------------------------------------------------------------

• Va reafirma els resultats de Mirzavec en la col·∙locació de les diferents H respecte la intereccio amb el DNA
1984: Es realitzen uns cristalls de la partícula nucli amb millor qualitat i que inclouen el DNA i ens permeten fer cristal·∙lografia. La imatge presenta una alta resolució per identificar el DNA pero en canvi una resolució mes baixa per veure el octàmer d’H. Per tant es un cristall que va servir per:
• Confirmar les mides: 11nm de diàmetre i 5,7nm d’alçada • Confirmar que el DNA esta per fora • Les zones protegides per l’acció de la DNAasa I no son com a resultat de que el
DNA es trobi protegit per les H sinó perquè aquestes son capaces de produir-‐li una distorsió en la estructura que fa que no pugui ser reconeguda per la DNAasa I
Moudrianakis: Es vana aconseguir un cristall perfectes de octàmers de H en les qual ses pot identificar perfectament la seva estructura. Cal descar que les cues no les podem veure ja que aquestes son molt flexibles.
• Disposició de les histones en l’octamer: Aquest esta format per una part central a la qual tenim el tetràmer de H3H4 i dos extrems formats per un dímer H2AH2B
• Motius estructurals de les H: Presenten un motiu anomenat motiu H que es basa en una hèlix-‐α curta loop una hèlix-‐α llarga un loop i una hèlix-‐α curta
• Interaccions de les H: Interaccionen entre elles mateixes i amb el soc petit del DNA. Les carregues positives del octàmer es col·∙loquen de tal manera que formen el que es coneix com a rampa helicoïdal i d’aquesta manera permet que el DNA adopti aquesta estructura al voltat del octàmer.
5. Empaquetament del DNA i replicació o transcripció
5.1 Introducció a la replicació amb presencia de nuclosomes Jose Maria Sogo: Estudia imatges de microscòpia electrònica de molècules de DNA que havien estat en un medi amb presencia de psoralent i a continuació fèiem una desnaturalització. El psoralent es capaç d’unir covalentment les dues cadenes com ja vam veure i per tant es capaç de que en condicions desnaturalitzant es conservi l’estructura de doble cadena, però en presencia de nucleosomes el psoralent no pot unir les dues cadenes.
JR.Daban BIOLOGIA MOLECULAR Figura 6.20
------------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.20
------------------------------------------------------------------------------------------------

Per tant si observem un DNA que contenia nucleosomes podrem veure que en les zones que hi havia nucleosomes hi veurem unes bombolles de ssDNA. Cal remarcar que aquest fet serà observable en el DNA que estigui nickat ja que el DNA CCC no es desnaturalitzarà. En els DNA que s’estaven produint una replicació es pot veure com les bombolles creades pels nucleosomes es troben immediatament després i abans de la forqueta de replicació. Conseqüentment ens indica que poden haver-‐hi diverses estratègies en vers la replicació i els nucleosomes
• Que el octàmer es dissocià just abans de que passi la polimerasa i es torna a associar just despres de que així pasat la polimerasa
• Que la polimerasa es capaç de replicar el DNA donant la volta a la hèlix que fa el DNA en el nucleosoma
• Que la polimerasa es capaç de replicar el DNA sense que es dissociï el octàmer però que sigui capaç de linealitzar-‐lo, es a dir, eliminar les volta i 1/3 que fa
5.2 Comprovació de la teoria Jackob: Per comprovar quina de la teoria es la que realment passa durant la replicació es va utilitzar un DNA unit a histones que es va fer replicar en un medi que contenia la polimerasa i un conjunt de H marcades radioactivament. Una vegada realitzada la replicació podien succeir dues coses:
• El octàmer estarà format per un conjunt histones no marcades i per tant vol dir que aquest octàmer no es dissocia com a conseqüència de la replicació, es a dir que es conservaven
• El octàmer estarà format per un conjunt de histones marcades i altres no marcades i per tant vol dir que aquest octàmer es dissocia i es torna a associar com a conseqüència de la replicació, es a dir que no es conservaven
El que es va observar va ser la segona opció per tant es va confirmar la teoria de que el octàmer es dissocià just abans de que passi la polimerasa i es torna a associar just després de que així passat la polimerasa
5.3 Mecanisme de associació i reassociació Daban: Utilitza la seqüencia del nucleosoma que ja havíem seqüenciat anteriorment. Aquest seqüencia s’ha l’hi ha inserit un promotor per la T7RNA pol que es una RNApol vírica i estarà unida a un nucleosoma. D’aquesta manera es podrà estudiar el procés de replicació o traducció en la forma mínima de plegament (tamplate cromatínic mínim). Aquesta seqüencia es coneguda com a NX1P7.
JR.Daban BIOLOGIA MOLECULAR Figura 6.24
------------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.26
------------------------------------------------------------------------------------------------

També utilitzaré un gel electroforètic anomenat de retardament o de nucloproteina que es capaç de analitzar tots els productes d’una transcripció mes la presencia de proteïnes i proteïna-‐DNA. Per tant tenim una banda pel DNA-‐pur, una banda pel RNA, una banda per el DNA unit a H i una banda que no entre en el gel i que correspon a un agregat de nucleosomes amb excés de H. Posteriorment es ban comparar les bandes que apareixien en presencia o no de la RNA polT7. El que es va veure era que sense la presencia de RNA pol tenim DNA no associat, DNA associat al nuclosoma i agregat de H,. Però en presencia de la RNApol es veu com ha aparegut una banda de mes corresponent a RNA, per tant es afirma que hi ha hagut transcripció a més a més veiem que a banda que pertant al DNA unit al nucloeoma s’ha fet mes prima i que la del DNA lliure i la del agregat de H s’ha fet mes gran i intensa. Per tant mentre que quan no hi havia RNA pol es mantenia un equilibri entre el DNA associat a histones i el DNA no associat a histones, la presencia de la RNA pol crea que el DNA associat a histones es dissoci però aquestes histones formaran un agregat que serà molt estable i per tant quan acabi la replicació no retornaran a formar DNA associat a histones. Conseqüentment la teoria que es va desenvolupar amb altres dades experimentals es que in vivo aquest fenomen d’agregació també s’ucceix però que el agregat no es tant estable com el de in vitro i per tant es dissocia ràpidament i això permet que es pugui tornar a formar ràpidament DNA associat a histones. Aquesta idea te molta lògica si pensem en les grans concentracions de DNA que hi han en els nuclis.
5.4 Com la polimerasa pot trencar el octamer Per tal de que la polimerasa pugui provocar la dissociació del octàmer es necessari que aquets actui com una pala i per això necessita:
• Punt d’ancoratge: Tant la DNApol en el procés de replicació com la RNApol en el procés de transcripció engloben un uns 15pb que ja aparellades. Aquestes bases permeten que la polimerasa no tiri cap enrere
• Energia: Cada vegada que s’uneix un nucleòtid es produeix l’alliberació d’un pirofosfat que permet donar l’energia suficient perquè la pol abansi una posicio cap endavant per atacar la següent base
JR.Daban BIOLOGIA MOLECULAR Figura 6.25
------------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.25
------------------------------------------------------------------------------------------------

Per tant diem que la pol fa una força motor.
6. Estructures d’ordre superior Definició: Son un conjunt d’estructures que es forment a partir de l’unio de diversos cromosomes i que permeten garantir el empaquetament
6.1 Histona H1 Es una proteïna que no es troba en el octàmer de histones i que sabem que s’uneix al DNA linker i permet que aquest pugui adoptar estructures d’ordres superiors S’ha pogut cristal·∙litzar la part globular de la H1 després de sotmetre la a una proteòlisi que fos capaç d’eliminar les dues cues (C-‐ter i N-‐ter). Mitjançant la difracció de raig X s’ha pogut determinar que aquesta molècula presenta un motiu hèlix-‐turn-‐helix que es molt recorrent en proteïnes de reconeixement de DNA ja que una de les hèlix (en el cas de la H1 es la hèlix III) es capaç d’inserir-‐se en el solc gran del DNA i realitzat ponts d’H amb les bases (en el cas de la H1 els ponts d’H seran inespecífics). Algunes de les proteïnes que tenen aques motiu són proteïna CAP (relacionada amb l’operon) que es capaç de reconèixer seqüencies especifiques de DNA i d’altres proteïnes d’interacció especifica, però cal remarcar que la H1 a grosso modo fa una interacció inespecífica.
6.2 Grau d’empaquetament Longitud del DNA en el interior de la cèl·∙lula: Tenint en compte que 3Gb podem determinar que:
36𝐺𝑏 ·1 · 10!𝑝𝑏1𝐺𝑏 ·
0,34𝑛𝑚1𝑝𝑏 ≈ 1 · 10!𝜇𝑚 = 1𝑚
Per tant tenim 1m de DNA que ha de fer entrar en el nucli i per això necessitem el empaquetament. Cal remarcar que hi han organismes que contenen 10m de DNA Packing Ratio: Es una formula per calcular i poder comparar el grau d’empaquetament
Per tant podem veure com el DNA es capaç d’empaquetar-‐se o de disminuir 10.000 la seva longitud. Si mirem quin es el grau d’empaquetament en el nucleosoma
𝐿𝑜𝑛𝑔𝑖𝑡𝑢𝑑 𝑑𝑒𝑙 𝐷𝑁𝐴 𝑒𝑛 𝑒𝑙 𝑛𝑢𝑐𝑙𝑒𝑜𝑠𝑜𝑚𝑎 = 200𝑝𝑏 ·0,34𝑛𝑚1𝑝𝑏 = 68𝑛𝑚

Per tant podem veure com amb els nucleosomes aconseguim empaquetar nomes 7 vegades el DNA i en realitat esta empaquetat 10.000 vegades, per tant es necessari que existeixin altres estructures d’empaquetament
6.3 Observació de les estructures d’ordre superior Cristal·∙lografia: No es pot formar cristalls perquè presenten una estructura que no es regular, molt heterogènia, amb un gran pm. Microscopi electrònic: Per tal de poder observar els diferents nivells estructurals es necessari que controli la concentració iònica del medi ja que aquestes sals son capaces de regular el empaquetament. Entre elles es troben el Na, K i Mg, aquest últim te un paper estructural molt important gracies a la seva divalència. D’aquesta manera:
• Més concentració de sals: Fibra de 10nm • Menys concentració de sals: Fibra de 30nm
Per tant a més concentració més grau d’empaquetament.
6.4 Nivells dels graus d’ordre superior Si mirem de menys empaquetament a mes empaquetament podem veure que:
• Fibra de 10 nm (diamentre del nucleosoma) o collaret de perles • Fibra de 30nm o solenoide • Llaçades • Cromosoma metafasic
6.5.1 Models de la fibra de 30nm Solenoide o plegament helicoïdal d’un sol origen: La fibra de 10nm s’estructura en forma de hèlix d’un sol origen en la qual els nucleosomes es col·∙loquen un al costat de l’altre i tenen un pas de rosca de 6 nucleosomes/volta. El DNA linker esta corbat al voltant de les partícules nucli i pasa d’un solenoide a un altre perquè com ja hem dit aquets estan contigus. Per tant aquesta fibra te un forat al mig Model cinta helicoïdal: S’observa un intermediari entre la fibra de 10nm i la solenoide que es una estrucrtura en zig-‐zag i per tant si aquesta forma una estructura helicoïdal el DNA linker quedarà paral·∙lel al eix de la helicoide. Per tant aquesta fibra te un forat al mig
JR.Daban BIOLOGIA MOLECULAR Figura 6.30
----------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.30
----------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.30
----------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.30
----------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.30
----------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.29
-----------------------------------------------------------------------------------------------

Model linker creuat: La helicoide es forma a partir de dues fibres, es a dir, de dos origen, i d’aquesta manera es com si en ves de tenir una fibra de 10nm tota en fila tinguéssim dues fibres de 10nm en paral·∙leles on el linker fa zig-‐zag i cadascuna d’aquestes fibres formes una hèlix. El DNA linker es trobarà justament al mig. Per tant aquesta fibra no te un forat al mig Model de super-‐beats: La fibra de 30nm no es continua formada per diverses agrupacions de nucleosoma que no segueixen cap ordre. El DNA linker uniria les diverses agrupacions Model del solenoide interdigitat: En grup del Daban va poder veure que la fibra de 30nm era molt mes compacte del que realment mostraven els altres models es a dir que tenen mes nucleosomes per unitat de longitud. 6.5.2 Cromosoma metafísic Leanli: Observa un cromosoma metafasic amb un medi d’aigua pura es a dir, amb una concentració salina igual a 0. S’observa com li surten petits “pels” en el cromosoma.
JR.Daban BIOLOGIA MOLECULAR Figura 6.30
----------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.30
----------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.30
----------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.30
----------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.31
----------------------------------------------------------------------------------------------
JR.D
aban BIO
LOG
IA MO
LEC
ULA
R
Figura 6.32
-----------------------------------------------------------------------------------------------

Posteriorment elimina les histones del medi i observa el cromosoma a més ampliació i va veure com les fibres estaven formant llaçades al voltant d’una estructura a al qual ell va anomenar scaffold. Per això ell va anomenar aquest model llaçades radials. La seva teoria era que el scaffold esta format per proteïnes no histoniques que serviran per organitza i fer de vestidor al cromosoma Aquest model es una teoria ja que per tal de poder-‐ho observar vàren tindre que utilitzar condicions desnaturalitzats i eliminar per complert les histones per tant no podem assegurar que això passi a nivell fisiològic Per aquest mateix motiu es va torna a repetir el experiment pero aquesta vegada en presencia d’histones i va poder reafirmar que existien unes llaçades al voltat d’una estructura que podia ser el scaffold. Aquestes llaçades es feien a partir de la fibra de 30nm Conexio nucli interfàsic-‐nucli metafasic: Es va fer una preparació de nuclis en el qual es van eliminar totes les proteïnes del interior del nucli i tot el material genètic. Es va observar per microscopi electronic i el que es va veure era una estructura que recordava molt el nucli. També es va poder observar que hi havien zones del DNA en el cromosoma que eren molt resistents a la degradació. Per tant la seva conclusió va ser que existia una matriu en el nucli interfàsic que seria la que posteriorment formarà el scaffold. Aquesta matriu ja presentava unides les diferents llaçades mitjançant seqüencies especifiques però aquestes es trobaven molt separades entre elles, però durant la mitosi quan el nucli desapareix aquesta matriu es va ajuntant i junt ara les diferents llaçades. Les sequencies d’unio a la matriu o al scaffold són:
• SAR: Unió a scaffold • MAR: Unió a matriu
Composició del scaffold: En el nucli la proteïna no histonica que s’hi troba en mes concentració es la topo II i una anomenada SMC. Per tant aquestes dues potser que
JR.Daban BIOLOGIA MOLECULAR Figura 6.33
----------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.33
----------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.33
----------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.35
----------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.36
----------------------------------------------------------------------------------------------

tinguin una doble funció i que durant la divisió cèl·∙lula siguin les que estiguin presents en el scaffold. Es sap que el scaffold pot contenir mes de 100 proteïnes diferents. Martio: Rebutja la teoria del model de les llaçades radials. Agafa un cromosoma pels dos extrems amb unes micropipetes molt petites i el tiba el màxim que pot. Posteriorment digereix amb nucleasa microcoal i veu com l’estructura no es manté recta sinó que es va corbant i finalment es trenca. Si hi hagués un scaffold això no podria passar
7. Model capes primes. Daban Definició: Els cromosomes metafísics s’organitzen en lamines de 6nm (alçada nucleosoma) apilades una al costat de l’altre i perpendiculars al eix de la cromàtide. Aquestes lamines estan formades per interdigitacions entre diferents capes ja que d’aquesta manera s’aconsegueix un alt grau d’empaquetament.
Aquestes lamines es van formant progressivament ja que primer s’omple tota una capa i després s’omple la següent capa i això fa que realment les capes estiguin connectades entre elles i formin un helicoide com pot ser una rampa d’un pàrquing Per tal de poder veure aquestes imatges es necessari aplicar unes condicions poc desnaturalitzants al cromosoma i després observar-‐los sense presencia de ions.
JR.Daban BIOLOGIA MOLECULAR Figura 6.37
----------------------------------------------------------------------------------------------
JR.Daban BIOLOGIA MOLECULAR Figura 6.37
----------------------------------------------------------------------------------------------BIOLOGIA
AVENÇOS
L'activitat genètica en els micronuclis: es potconsiderar realment perdut el DNAmicronuclear?
Aquesta recerca ha estudiat la naturalesa delsmicronuclis, estructures produïde per irregularitatsen el procés de divisió cel·lular. Els resultatsindiquen que la pròpia naturalesa dels micronuclisés la responsable de la destinació que pateixen, elquè suposa un avenç important en el coneixementd'aquestes estructures utilitzades per detectarinestabilitats cromosòmiques.[+]
AVENÇOS
Un fàrmac redueix en ratolins la por causadaper traumes previs
Haver viscut una experiència traumàtica afavoreix lapersistència de la por associada a un estímulaversiu, una por condicionada que es pot alleujar enratolins amb una sola dosi de 7,8-dihidroxiflavona,un derivat flavonoide que potencia nousaprenentatges emocionals. El fàrmac podria sereficaç per tractar trastorns d'estrès post traumàtic,de pànic i fòbies en persones.[+]
AVENÇOS
La virulència del Streptococcus suis queda aldescobert
Investigadors de la UAB, amb col·laboració amb elGREMIP de Quebec, han aconseguit produir unsmutants de Streptococcus suis amb unadesregularització en el transport de zinc i de ferroque han mostrat una capacitat infecciosa moltatenuada. Aquesta recerca obre les portes per aldesenvolupament de vacunes efectives contraaquest patogen.[+]
AVENÇOS
Lluitar contra els virus a les granges seguint laseva evolució filogenètica
La irrupció de l'anàlisi genètica com a mètode dediagnosi de malalties ha suposat un avenç importantdel que la indústria ramadera s'està beneficiant.L'estudi de les seqüències de nucleòtids dels viruspermet fer reconstruccions filogenètiques quepermetran deduir característiques d'aquests agentspatògens, podent seguir l'evolució del virus i traçarl'origen d'un brot.[+]
A A A Contrast +/- Castellano | English CERCA
ACTIVITATS TESIS ENTREVISTES AVENÇOS A FONS
09/2010 - Nou model estructural en capes primes per al cromosomametafàsic
Experiments realitzats fent servir molt diverses tècniques de microscòpia hanpermès als investigadors del Laboratori de Cromatina de la UAB descobrir que,durant la divisió cel·lular, els cromosomes tenen el DNA empaquetat dins d’unesestructures planes que estan formades de moltes capes molt primes. Aquestesestructures planes estan apilades, omplen tot el volum dels cromosomes iprobablement estan orientades perpendicularment respecte a l’eix central de lescromàtides.
Referències
"Dense chromatin plates in metaphase chromosomes". I. Gállego, P. Castro-Hartmann,J.M. Caravaca, S. Caño i J.R. Daban. European Biophysics Journal, 38 (2009) 503-522.
"Irregular orientation of nucleosomes in the well-defined chromatin plates of metaphasechromosomes". P. Castro-Hartmann, M. Milla i J.R. Daban. Biochemistry, 49 (2010)4043-4050.
En un treball previ del nostre laboratori varem observar plaques formades per vàriescapes a l’entorn de cromosomes humans parcialment desnaturalitzats en presència deconcentracions iòniques properes a les que existeixen en la metafase. Aquesta inesperadaobservació ens ha portat a realitzar una extensa investigació sobre aquestes estructureslaminars. Els nostres resultats de microscòpia electrònica de transmissió (TEM) mostrenque les plaques també es poden veure en els cromosomes metafàsics de pollastre, unaespècie evolutivament molt allunyada dels humans. Hem observat que la simple diluciódels cromosomes amb solucions hiposmòtiques pot transformar tota la cromàtida enplaques esteses multilaminars. A partir d’aquests resultats hem suggerit que elscromosomes condensats estan formats per plaques de cromatina apilades i perpendicularsa l’eix de les cromàtides. Els cromosomes natius són molt compactes; per això les plaquestan sols es poden veure quan els cromosomes estan parcialment desnaturalitzats.
Hem emprat microscòpia de força atòmica (AFM) per obtenir imatges i per investigar lespropietats mecàniques de les plaques directament en medi aquós. Les plaques tenen ungruix petit (aproximadament 6 nm) però són compactes i resistents a la penetració de lasonda del microscopi; es necessita una força de 5 nN per a la completa penetració de laplaca amb la punta de la sonda. La incubació amb medis de molt baixa concentració iònicaprovoca l’emanació de fibres de cromatina exclusivament a la perifèria de les plaques.Aquest estudis demostren que el filament de cromatina es troba fortament lligat a leszones internes de les plaques.
Hem investigat l’estructura de les plaques mitjançant microscòpia electrònica de rastreig(SEM), criomicroscòpia electrònica i tomografia electrònica. Les reconstruccionstridimensionals obtingudes en els estudis de tomografia indiquen que les plaques tenenuna superfície llisa, sense estructures repetitives que puguin ésser interpretades com aorganitzacions regulars dels nucleosomes.
També hem estudiat els cromosomes dins de cèl·lules metafàsiques en solució aquosamitjançant microscòpia de polarització. Els resultats mostren que els cromosomes no sónbirefringents en condicions iòniques similars a les emprades amb les plaques. Aquestaobservació és incompatible amb l’existència de columnes paral·leles de nucleosomes dinsels cromosomes.
No hem detectat doncs cap orientació regular dels nucleosomes, però els nostres resultatsindiquen que la cromatina en els cromosomes natius està organitzada formant plaquesben definides. Per poder justificar el petit gruix que hem observat per les plaques, hemsuggerit que molt probablement hi ha una interdigitació de les successives capes. Pensemque la compactació aconseguida és necessària per la transferència segura del DNAgenòmic a les cèl·lules filles durant la mitosi. El model de les plaques primes aporta unainterpretació completament nova de l’estructura del cromosoma metafàsic. El nostremodel dóna les bases físiques per poder explicar de manera senzilla la formació de lesbandes dels cromosomes que són estudiades en les anàlisis citogenètiques.
Les imatges de TEM, SEM, i de criomicroscòpia i tomografia electrònica han estatobtingudes al Servei de Microscòpia de la UAB, les imatges d’AFM i els espectres de forçaals Serveis Científico-Tècnics de la UB, i les imatges de microscòpia de polarització alServei de Tractament i Anàlisi d’Imatges de la UAB.
Nou model estructural en capes primes per al cromosoma metafàsic U... http://www.uab.cat/servlet/Satellite?cid=1096481464166&pagename...
1 de 2 12/04/2011 19:49
BIOLOGIA
AVENÇOS
L'activitat genètica en els micronuclis: es potconsiderar realment perdut el DNAmicronuclear?
Aquesta recerca ha estudiat la naturalesa delsmicronuclis, estructures produïde per irregularitatsen el procés de divisió cel·lular. Els resultatsindiquen que la pròpia naturalesa dels micronuclisés la responsable de la destinació que pateixen, elquè suposa un avenç important en el coneixementd'aquestes estructures utilitzades per detectarinestabilitats cromosòmiques.[+]
AVENÇOS
Un fàrmac redueix en ratolins la por causadaper traumes previs
Haver viscut una experiència traumàtica afavoreix lapersistència de la por associada a un estímulaversiu, una por condicionada que es pot alleujar enratolins amb una sola dosi de 7,8-dihidroxiflavona,un derivat flavonoide que potencia nousaprenentatges emocionals. El fàrmac podria sereficaç per tractar trastorns d'estrès post traumàtic,de pànic i fòbies en persones.[+]
AVENÇOS
La virulència del Streptococcus suis queda aldescobert
Investigadors de la UAB, amb col·laboració amb elGREMIP de Quebec, han aconseguit produir unsmutants de Streptococcus suis amb unadesregularització en el transport de zinc i de ferroque han mostrat una capacitat infecciosa moltatenuada. Aquesta recerca obre les portes per aldesenvolupament de vacunes efectives contraaquest patogen.[+]
AVENÇOS
Lluitar contra els virus a les granges seguint laseva evolució filogenètica
La irrupció de l'anàlisi genètica com a mètode dediagnosi de malalties ha suposat un avenç importantdel que la indústria ramadera s'està beneficiant.L'estudi de les seqüències de nucleòtids dels viruspermet fer reconstruccions filogenètiques quepermetran deduir característiques d'aquests agentspatògens, podent seguir l'evolució del virus i traçarl'origen d'un brot.[+]
A A A Contrast +/- Castellano | English CERCA
ACTIVITATS TESIS ENTREVISTES AVENÇOS A FONS
09/2010 - Nou model estructural en capes primes per al cromosomametafàsic
Experiments realitzats fent servir molt diverses tècniques de microscòpia hanpermès als investigadors del Laboratori de Cromatina de la UAB descobrir que,durant la divisió cel·lular, els cromosomes tenen el DNA empaquetat dins d’unesestructures planes que estan formades de moltes capes molt primes. Aquestesestructures planes estan apilades, omplen tot el volum dels cromosomes iprobablement estan orientades perpendicularment respecte a l’eix central de lescromàtides.
Referències
"Dense chromatin plates in metaphase chromosomes". I. Gállego, P. Castro-Hartmann,J.M. Caravaca, S. Caño i J.R. Daban. European Biophysics Journal, 38 (2009) 503-522.
"Irregular orientation of nucleosomes in the well-defined chromatin plates of metaphasechromosomes". P. Castro-Hartmann, M. Milla i J.R. Daban. Biochemistry, 49 (2010)4043-4050.
En un treball previ del nostre laboratori varem observar plaques formades per vàriescapes a l’entorn de cromosomes humans parcialment desnaturalitzats en presència deconcentracions iòniques properes a les que existeixen en la metafase. Aquesta inesperadaobservació ens ha portat a realitzar una extensa investigació sobre aquestes estructureslaminars. Els nostres resultats de microscòpia electrònica de transmissió (TEM) mostrenque les plaques també es poden veure en els cromosomes metafàsics de pollastre, unaespècie evolutivament molt allunyada dels humans. Hem observat que la simple diluciódels cromosomes amb solucions hiposmòtiques pot transformar tota la cromàtida enplaques esteses multilaminars. A partir d’aquests resultats hem suggerit que elscromosomes condensats estan formats per plaques de cromatina apilades i perpendicularsa l’eix de les cromàtides. Els cromosomes natius són molt compactes; per això les plaquestan sols es poden veure quan els cromosomes estan parcialment desnaturalitzats.
Hem emprat microscòpia de força atòmica (AFM) per obtenir imatges i per investigar lespropietats mecàniques de les plaques directament en medi aquós. Les plaques tenen ungruix petit (aproximadament 6 nm) però són compactes i resistents a la penetració de lasonda del microscopi; es necessita una força de 5 nN per a la completa penetració de laplaca amb la punta de la sonda. La incubació amb medis de molt baixa concentració iònicaprovoca l’emanació de fibres de cromatina exclusivament a la perifèria de les plaques.Aquest estudis demostren que el filament de cromatina es troba fortament lligat a leszones internes de les plaques.
Hem investigat l’estructura de les plaques mitjançant microscòpia electrònica de rastreig(SEM), criomicroscòpia electrònica i tomografia electrònica. Les reconstruccionstridimensionals obtingudes en els estudis de tomografia indiquen que les plaques tenenuna superfície llisa, sense estructures repetitives que puguin ésser interpretades com aorganitzacions regulars dels nucleosomes.
També hem estudiat els cromosomes dins de cèl·lules metafàsiques en solució aquosamitjançant microscòpia de polarització. Els resultats mostren que els cromosomes no sónbirefringents en condicions iòniques similars a les emprades amb les plaques. Aquestaobservació és incompatible amb l’existència de columnes paral·leles de nucleosomes dinsels cromosomes.
No hem detectat doncs cap orientació regular dels nucleosomes, però els nostres resultatsindiquen que la cromatina en els cromosomes natius està organitzada formant plaquesben definides. Per poder justificar el petit gruix que hem observat per les plaques, hemsuggerit que molt probablement hi ha una interdigitació de les successives capes. Pensemque la compactació aconseguida és necessària per la transferència segura del DNAgenòmic a les cèl·lules filles durant la mitosi. El model de les plaques primes aporta unainterpretació completament nova de l’estructura del cromosoma metafàsic. El nostremodel dóna les bases físiques per poder explicar de manera senzilla la formació de lesbandes dels cromosomes que són estudiades en les anàlisis citogenètiques.
Les imatges de TEM, SEM, i de criomicroscòpia i tomografia electrònica han estatobtingudes al Servei de Microscòpia de la UAB, les imatges d’AFM i els espectres de forçaals Serveis Científico-Tècnics de la UB, i les imatges de microscòpia de polarització alServei de Tractament i Anàlisi d’Imatges de la UAB.
Nou model estructural en capes primes per al cromosoma metafàsic U... http://www.uab.cat/servlet/Satellite?cid=1096481464166&pagename...
1 de 2 12/04/2011 19:49